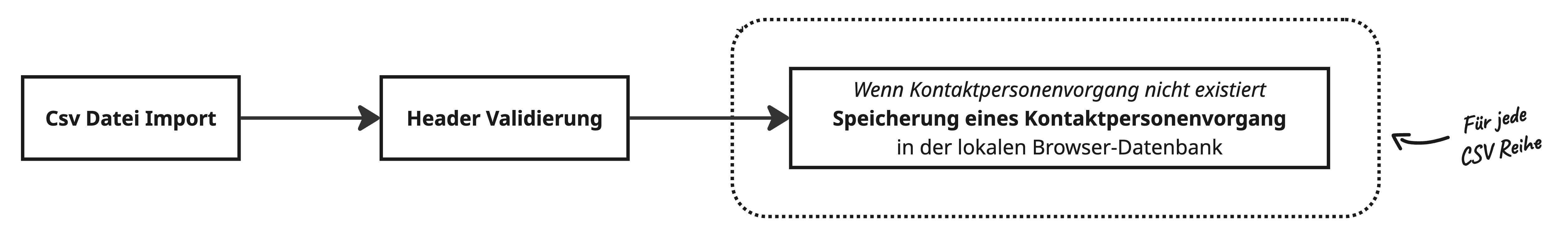
Datenverarbeitung
Ausbruchsanalysen basieren auf Minimum Spanning Tree (MST)-Visualisierungen von Infektionsfällen, die durch genetische Distanzen oder Kontaktverfolgungsereignisse verbunden sind. Diese MSTs sind auf einen importierten Datensatz angewiesen, der aus Fällen, sequenzierten Proben und Kontaktinformationen besteht.
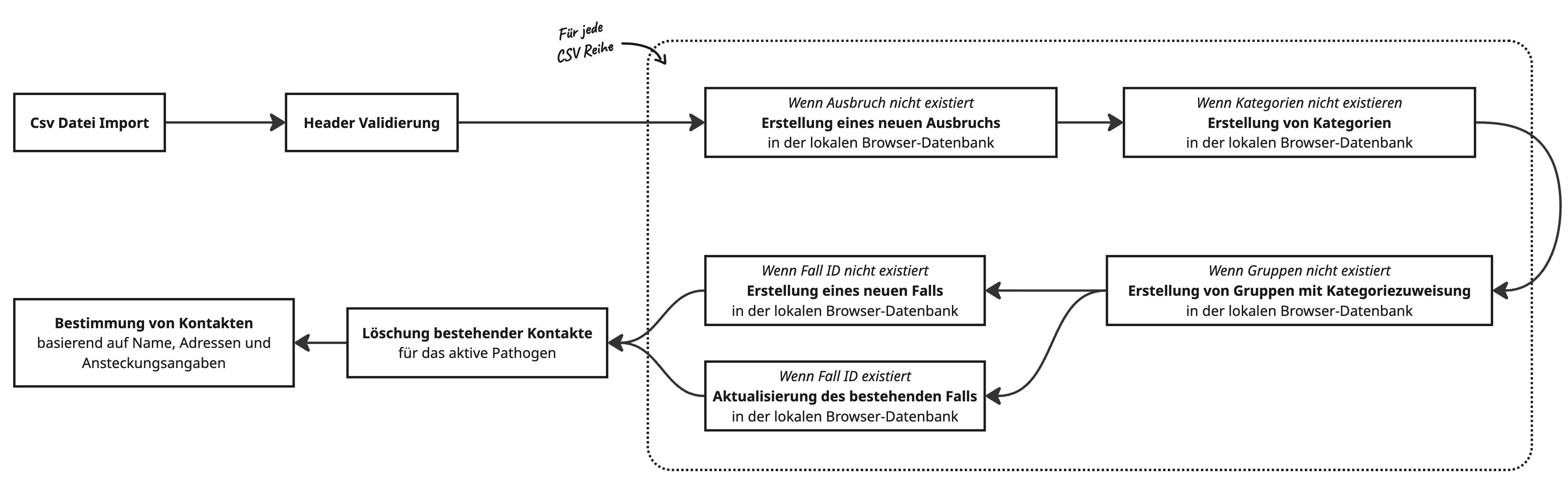
Datei-Importe
Falldaten
Fälle sind registrierte Infektionsmeldungen von den Gesundheitsbehörden. Diese werden mit der vom RKI entwickelten SurvNet-Software erfasst. Personenbezogene Daten werden ausschließlich clientseitig gehandhabt und gespeichert. Adressen und Namen werden verwendet, um Kontaktkanten zwischen Fällen zu erstellen, da es eine wertvolle Information ist, wenn Fälle an derselben Adresse leben (Wohngemeinschaften, Seniorenheime, ...) oder denselben Nachnamen haben (potenzielle Familienmitglieder).
Für den Import von Falldaten aus SurvNet wurde eine CSV-Struktur mit relevanten Feldern erstellt:
| Field | Naming options | Description | Required |
|---|---|---|---|
| Case id | Fall ID, Aktenzeichen | Unique ID of the case | ✅ |
| Registration date | Registrierungsdatum, Meldedatum | Date of registration in SurvNets | ✅ |
| Sequence id | Sequenz ID | Fasta ID of the corresponding sequence | |
| Outbreak | Ausbruch, AusbruchInfo_InternalName, AusbruchInfo_NameGA, AusbruchInfo_NameLS, AusbruchInfo_NameRKI,AusbruchInfo_GuidRecord, AusbruchInfo_Aktenzeichen, AusbruchInfo_InterneRef | Suspected outbreak association | |
| Infected by | Angesteckt bei, AngestecktBei | Unique ID of a case that was given as the origin of the infection | |
| Firstname | Vorname, PersonVorname | First name of the person associated with the case | |
| Lastname | Nachname, PersonFamilienname | Last name of the person associated with the case | |
| City | Ort, PersonOrt | Place of residence of the person associated with the case | |
| Zip code | PLZ, PersonPLZ | Zip code of the person associated with the case | |
| Street | Straße, PersonStrasse | Street of the person associated with the case | |
| Flexible category | Kategorie:{category_name} | Flexible category for further differentiation |
Verarbeitung von Fällen
Sequenzdaten
Zwischen allen importierten Sequenzen werden genetische Distanzen berechnet. Hierzu wird zunächst eine komprimierte Repräsentation der Sequenzen auf Basis der vorliegenden Mutationen erzeugt. Im Falle von Viren verwendet GENTRAIN Nextclade, während chewBBACA für die Bestimmung vorliegender Allele eingesetzt wird. Auf Basis der Mutationen können anschließend genetische Distanzen zwischen Genomsequenzen berechnet werden, die anschließend in einer Distanzmatrix zusammengefasst werden. Diese Distanzmatrix ermöglicht es, einen Minimum Spanning Tree für die Fälle auf Basis der genetischen Distanz zu erstellen.
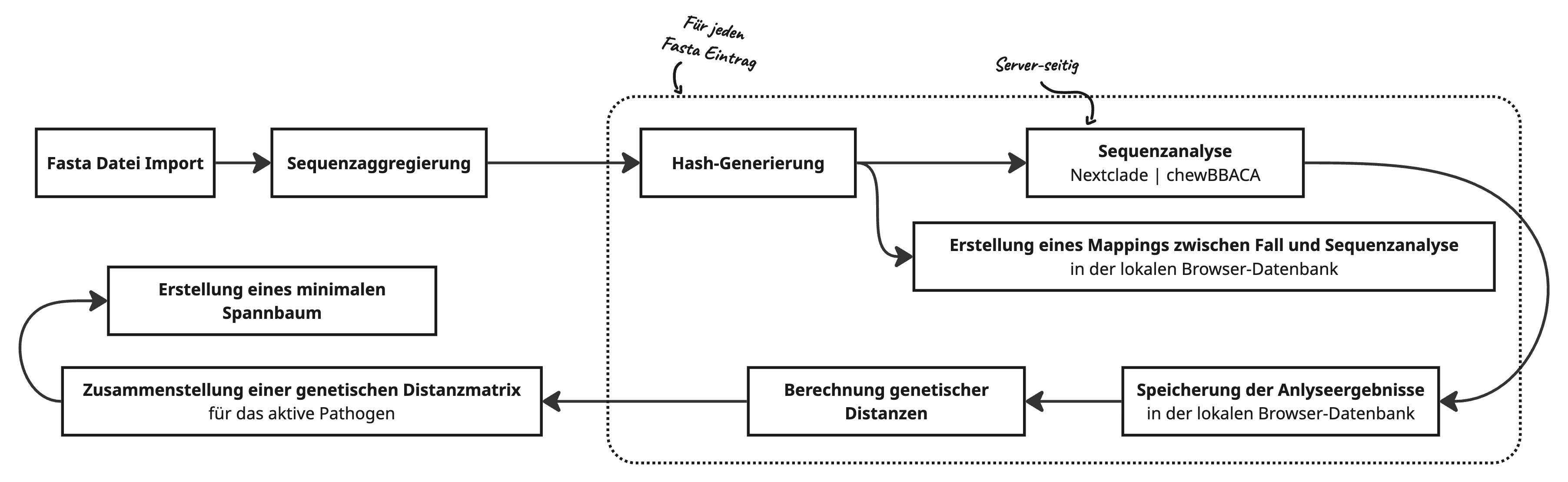
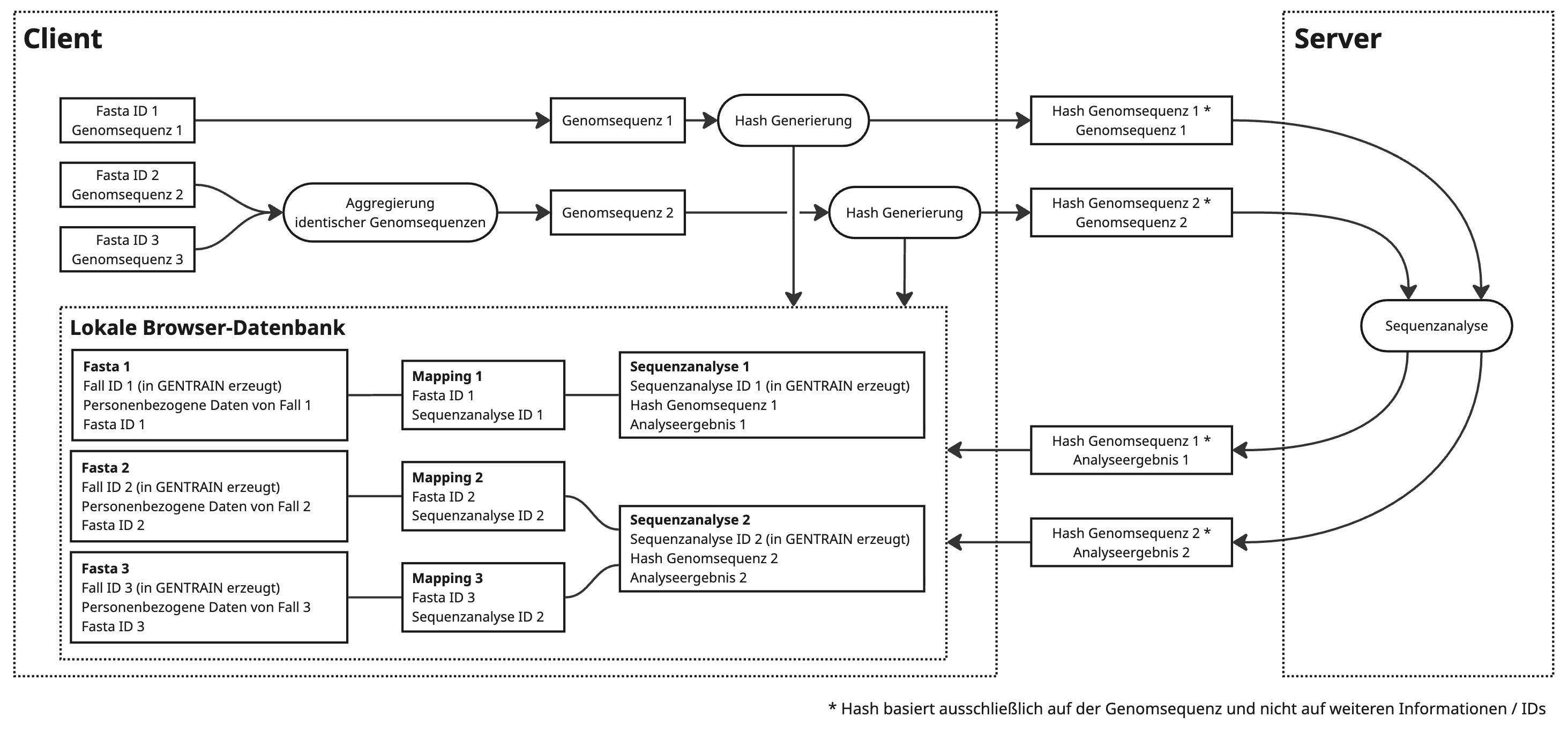
Aggregierung identischer Sequenzen
Fälle können identische Genomsequenzen aufweisen. Um Speicherplatz zu minimieren und eine serverseitige Anonymisierung zu gewährleisten, werden identische Sequenzdaten in GENTRAIN zu einer Sequenzanalyse aggregiert. Zur clientseitigen Zuordnung zwischen Fällen und Ergebnissen der Sequenzanalyse werden die gehashten Sequenzen verwendet.
WebSocket Kommunikation
Da die Sequenzanalyse einige Minuten in Anspruch nehmen kann, wird sie in Form von Jobs in einer Redis-Queue abgearbeitet. Für die Kommunikation zwischen Client und Server wurde daher ein persistenter Kanal mittels WebSockets eingerichtet, um einen zuverlässigen Informationsaustausch zu gewährleisten. Der WebSocket-Server wurde mit Flask SocketIO implementiert, die WebSocket-Clients mit Socket.io.
- Erstellung eines WebSocket-Raumes für den Analyse-Prozess
- Chunking der Sequenzdaten
- Übermittelung der Sequenzdaten-Chunks (mit Sequenz-Hashes zur Identifizierung) an den WebSocket-Raum
- Sobald alle Chunks übermittelt wurden: Enqueueing des Sequenzanalyse-Jobs in die Redis Queue
- Nach Abschluss des Sequenzanalyse-Jobs: Übermittelung des Sequenzanalyse-Ergebnisses an den WebSocket-Client
Sequenzanalyse
Um die genetischen Abstände zwischen viralen und bakteriellen Genomen zu berechnen, analysieren wir genetische Sequenzen anhand ihres Referenzgenoms. Auf diese Weise gelingt es uns, eine minimierte Datenstruktur ohne nennenswerten Informationsverlust beizubehalten. Da virale Sequenzen in der Regel vollständig verfügbar sind und bakterielle Sequenzen meist zu Assemblies verarbeitet werden, kommen unterschiedliche Analysetechniken zum Einsatz.
Virale Sequenzanalyse
Für virale Sequenzen bestimmen wir Mutationen auf der Grundlage des entsprechenden Referenzgenoms, was mit Hilfe der Nextclade CLI erfolgt. Nextclade liefert Mutationsobjekte, die aus SNPs, Insertionen, Deletionen, Ns und Nicht-ACGTN-Zeichen bestehen. Diese Mutationsobjekte ermöglichen es uns, genetische Distanzen zu berechnen, ohne die vollständige Sequenz zu speichern. Alle Sequenzen in einer Fasta-Datei werden gleichzeitig als Teil eines einzigen Auftrags analysiert. Der Fasta-Inhalt wird anonymisiert, indem Sequenzen während des gesamten Analyseprozesses sowohl auf Client- als auch auf Serverseite durch Hash-Sequenzen ersetzt werden.
Nextclade liefert eine Reihe von Informationen zu jeder analysierten Probe. Diese Informationen können in verschiedenen Dateiformaten gespeichert werden, in unserem Fall erhalten wir ein JSON-Objekt. Dieses Objekt enthält Informationen über die erkannte Klade, Qualitätsmaße der Sequenzen und Mutationsinformationen. Erkannte Substitutionen, Insertionen, Deletionen, fehlende Zeichen und Nicht-ACGTNs der Sequenz werden im Browser des Benutzers gespeichert. Diese Informationen ermöglichen es uns, Sequenzen unter Berücksichtigung der Ausrichtung der Sequenzen zu rekonstruieren, ohne die gesamte Zeichenfolge zu erhalten.
{
substitutions: [
{pos: 209, refNuc: 'G', qryNuc: 'T'},
{pos: 240, refNuc: 'C', qryNuc: 'T'},
{pos: 3036, refNuc: 'C', qryNuc: 'T'},
...
{pos: 29741, refNuc: 'G', qryNuc: 'T'}
],
insertions: [
{pos: 18099, qryNuc: 'TCG'},
{pos: 29741, qryNuc: 'ACGT'}
],
deletions: [
{
range: {begin: 28247, end: 28253}
}
],
missings: [
{
character: 'N',
range: {begin: 0, end: 54}
},
{
character: 'N',
range: {begin: 6839, end: 6840}
}
],
nonACGTNs: [
{
character: 'Y',
range: {begin: 4504, end: 4505}
},
]
}
Es kann vorkommen, dass eine Sequenzierungstechnologie ein Nukleotid an einer bestimmten Position nicht eindeutig bestimmen kann. In diesem Fall wird ein mehrdeutiges Symbol gesetzt, das für alle möglichen Nukleotide an dieser Position stehen kann. Sind alle vier Nukleotide möglich, wird beispielsweise ein „N” verwendet. Diese mehrdeutigen Symbole sind in der IUPAC-Nomenklatur definiert.
{
"A": ["A"],
"C": ["C"],
"G": ["G"],
"T": ["T"],
"U": ["U"],
"M": ["A", "C"],
"R": ["A", "G"],
"S": ["C", "G"],
"W": ["A", "T"],
"Y": ["C", "T"],
"K": ["G", "T"],
"V": ["A", "C", "G"],
"H": ["A", "C", "T"],
"D": ["A", "G", "T"],
"B": ["C", "G", "T"],
"N": ["A", "C", "G", "T"],
"X": ["A", "C", "G", "T"]
}
Bakterielle Sequenzanalyse
Bakterielle Sequenzen werden mit chewBACCA analysiert. Laut eigener Dokumentation ist „chewBBACA eine Software-Suite zur Erstellung und Auswertung von Schemata und Ergebnissen für die MultiLocus-Sequenztypisierung (cg/wgMLST) des Kerngenoms und des gesamten Genoms“. Für Gentrain verwenden wir den AlleleCall-Dienst von chewBACCA , der Zuordnungen zwischen jedem Referenzgen und dem entsprechenden Allel in den Sequenzen bereitstellt. Basierend auf diesen Zuordnungen berechnen wir dann die genetischen Distanzen, indem wir zwischen den Allelsätzen zweier Sequenzen unterscheiden. Jede bakterielle Sequenzassemblierung wird in einer separaten Fasta-Datei bereitgestellt. Daher ermöglicht der Import bakterieller Sequenzen das gleichzeitige Hochladen mehrerer Dateien. Die Dateinamen müssen mit den Fasta-IDs übereinstimmen, die mit den hochgeladenen Falldaten verknüpft sind. Diese Fasta-IDs werden vor der Kommunikation mit dem Server pseudonymisiert und ausschließlich auf der Client-Seite gespeichert.
Die bakterielle Sequenzanalyse führt zu einer Zuordnung zwischen dem Referenzgen und der Allelsequenz der entsprechenden Probe (Gen-ID: Allelsequenz). Diese Allelsequenzen werden ebenfalls gehasht, um die Sequenzlänge zu minimieren und die Schemainabhängigkeit sicherzustellen.
{
SAUR0001: "d3627b0e335350fc61d130d50e6516b2",
SAUR0002: "34d94e7230113031e160b5880f0ed5af",
SAUR0003: "ddf68eaae82c14021c4f44f73ac0789f",
...
SAUR3016: "e25053695fa8a872d478c73aba78c26a"
}
Distanzbestimmung
Zwischen allen Fällen des aktiven Pathogens, für die ein Sequenzanalyse-Ergebnis vorliegt, können genetische Distanzen berechnet werden. Je nachdem, ob es sich um ein virales oder bakterielles Pathogen handelt, werden diese unterschiedlich bestimmt.
Virale Distanzbestimmung
Die genetischen Distanzen werden paarweise berechnet. Hierzu werden auf Basis der Nextclade-Analyseergebnisse und des Referenzgenoms vollständige Sequenzen rekonstruiert. Dabei werden abhängig von der gefundenen Mutation an einer Position des Referenzgenoms folgende Regeln befolgt.
| Mutationstyp | Regel zur Sequenzrekonstruktion |
|---|---|
| Substitution | Das ersetzende Nukleotid wird zu der Sequenz hinzugefügt, die an der aktuellen Referenzposition eine Subsitution aufweist. Auch mehrdeutige Symbole aus der IUPAC-Nomenklatur (inklusive Ns) werden als Subsitutions behandelt. |
| Insertion | Die eingefügten Nukleotide werden an die Sequenzen angehängt, die an der aktuellen Referenzposition eine Insertion aufweisen. Falls beide Sequenzen an der aktuellen Referenzposition eine Insertion enthalten, werden sie aligned, um die Überlappung beider Insertionen zu identifizieren. Für das Alignment wird der PairwiseAligner des Biopython-Pakets verwendet. Dieses Paket verwendet den Smith-Waterman-Algorithmus, einen weithin anerkannten Ansatz zur Ermittlung optimaler Alignments in Genomsequenzen. Wenn eine Sequenz an der aktuellen Position keine Insertion aufweist, werden die eingefügten Positionen mit Gaps gefüllt. |
| Deletion | Jeder Sequenz, die an der aktuellen Referenzposition eine Deletion aufweist, wird eine Lücke hinzugefügt. Wenn beide Sequenzen eine Deletion aufweisen, wird die Referenzposition vollständig übersprungen und den rekonstruierten Sequenzen nicht hinzugefügt. |
| Keine Mutation | Das Referenznukleotid für die aktuelle Position wird zu den Sequenzen hinzugefügt, die an der aktuellen Referenzposition keine Mutation aufweisen. |
Im nächsten Schritt werden die Symbole jeder einzelnen Position des Referenzgenoms mit den entsprechenden Positionen in den beiden rekonstruierten Sequenzen verglichen. Liegen für eine Position des Referenzgenoms unterschiedliche Symbole vor, wird die Distanz je nach den vorliegenden Symbolen inkrementell erhöht – oder auch nicht. Hierzu werden folgende Regeln befolgt.
| Symbol | Regel zur Distanzbestimmung |
|---|---|
| Missings (N) | Wenn eine der Sequenzen an der aktuellen Ausrichtungsposition N aufweist, wird der genetische Abstand nicht erhöht, um falsch-negative Ergebnisse zu vermeiden. |
| Andere mehrdeutige Symbole | Wenn eine Sequenz an der aktuellen Ausrichtungsposition eine Lücke aufweist, erhöht sich der genetische Abstand nur dann, wenn diese Sequenz an der vorherigen Ausrichtungsposition keine Lücke aufwies. |
| Gaps | Der genetische Abstand wird nur dann erhöht, wenn das Symbol an der aktuellen Ausrichtungsposition das Nukleotid der anderen Sequenz an der aktuellen nach der IUPAC-Nomenklatur Ausrichtungsposition nicht darstellen kann. |
Bakterielle Distanzbestimmung
Bei bakteriellen Proben ist dieser Vorgang recht trivial, da nur die Allel-Hashes pro Gen verglichen werden müssen. Unterschiedliche Allel-Hashes führen zu einer Distanzinkrementierung von 1. Der Distanzwert wird nicht erhöht, wenn das Allel einer Probe von chewBACCA nicht bestimmt werden konnte, um falsch positive Distanzimkrementierungen zu vermeiden.
Kontaktpersonendaten
Kontaktpersonen-Vorgänge liefern Informationen darüber, welche Fälle miteinander in Kontakt standen. Diese Informationen erweitern die Kontaktinformationen, die wir aus Adressen und Namen extrahieren. Der Import von Kontaktpersonen führt zu Kontaktkanten, die als 'Contact person' bezeichnet werden.
| Field | Naming options | Description | Required |
|---|---|---|---|
| Fall-ID 1 | Fall ID 1 | Eindeutige ID des ersten Falls des Kontaktpersonen-Vorgangs | ✅ |
| Fall-ID 2 | Fall ID 2 | Eindeutige ID des zweiten Falls des Kontaktpersonen-Vorgangs | ✅ |